|
Salmonelosis en lechones destetados de granjas de traspatio en Valledupar, Colombia*
ARTÍCULO DE INVESTIGACIÓN
Karelys Payares-Ramírez 1
1 Microbióloga, Joven Investigador, Universidad Popular del Cesar 2 Zootecnista, Docente Programa Medicina Veterinaria y Zootecnia Universidad de Santander 3 Microbióloga, Universidad Popular del Cesar 4 Microbiólogo, Docente Programa Microbiologia, Universidad Popular del Cesar
5 Microbióloga, Docente Programa Microbiologia, Universidad Popular del Cesar
This email address is being protected from spambots. You need JavaScript enabled to view it.
Recibido: 27 de agosto de 2018, Aprobado: 25 de octubre de 2018 y Actualizado: 28 de diciembre de 2018
DOI: 10.17151/vetzo.2019.13.1.4
RESUMEN. Introducción: La cría de cerdos en la costa Atlántica Colombiana se caracteriza por producciones de traspatio sin tecnificar y con protocolos de alimentación y sanidad deficientes. Objetivo: Determinar la presencia de Salmonella spp. en lechones destetados de granjas de traspatio en la ciudad de Valledupar (Colombia). Métodos: Se tomaron muestras en 90 lechones de nueve granjas de traspatio con sintomatología clínica aparente de salmonelosis. Las muestras se procesaron mediante el protocolo de aislamiento e identificación de Salmonella de la Organización Internacional de Epizootias. La confirmación de la especie se realizó mediante el equipo automatizado MicroScan Walkaway 96. Resultados: Se detectó la presencia de Salmonella spp. en el 3,3% de las muestras analizadas, en tres granjas de las nueve muestreadas. Además, se presentaron dos muestras positivas a Proteus mirabilis y Klebsiella pneumoniae. Conclusiones: El estudio demuestra el riesgo de diseminación de patógenos bacterianos que no solo afectan la salud animal, debido a las pérdidas económicas por muertes de lechones a causa de diarreas, sino al potencial zoonótico de estos microorganismos, causantes de problemas digestivos y urinarios en consumidores de carne de cerdo procedente de este tipo de producciones.
Palabras clave: cerdos, Klebsiella, Microscan, Proteus, Salmonella
Salmonellosis in weaned piglets from backyard farms in Valledupar, Colombia
ABSTRACT. Introduction: Pig farming on the Colombian Atlantic coast is characterized by backyard productions without technification and with both poor food and health management. Aims: To determine the presence of Salmonella spp. in weaned piglets from backyard farms in the city of Valledupar (Colombia). Methods: Samples were taken in 90 piglets with apparent clinical symptoms of salmonellosis from nine backyard farms. The samples were processed by the Salmonella isolation and identification protocol of the International Organization for Animal Health. Confirmation of the species was carried out using the MicroScan Walkaway 96 automated equipment. Results: Salmonella spp. was detected in 3.3% of the analyzed samples, in three of the nine farms sampled. In addition, two positive samples were detected to Proteus mirabilis and Klebsiella pneumoniae. Conclusions: The study demonstrates the risk of dissemination of bacterial pathogens that not only affect animal health, due to economic losses due to deaths of piglets due to diarrhea, but also to the zoonotic potential of these microorganisms, which cause digestive and urinary problems in consumers of pork from this type of production.
Key words: Klebsiella, Microscan, pigs, Proteus, Salmonella
Introducción
Las enfermedades del tracto gastrointestinal en cerdos casi siempre traen consigo diarreas, las cuales tienen lugar a causa de la inflamación del tracto intestinal, o debido a la interrupción de procesos de absorción y secreción de las células epiteliales del tracto digestivo (Bustos & Segura, 2005). El destete de los lechones constituye un período crítico en la producción porcina, debido a que son sometidos a mucho estrés y cambios en su inmunidad por la lactancia. Muchos patógenos colonizan el tracto gastrointestinal, como es el caso de Escherichia coli enterotoxigénica (ETEC), Clostridium perfringens, Salmonella spp. y rotavirus, los cuales están vinculados a las pérdidas en la producción, reducción en la tasa de crecimiento e incluso la muerte del animal (Vásquez, 2013). La salmonelosis puede afectar a todas las especies de animales domésticos; los más susceptibles son los animales jóvenes, en estado de gestación o lactantes, aunque algunos pueden estar infectados sin manifestar la enfermedad clínica, en especial los cerdos y las aves, quienes constituyen un factor clave en la difusión de la enfermedad entre explotaciones y como fuentes de contaminación alimentaria y de infección humana. (Organización de las Naciones Unidas para la Agricultura y la Alimentación [FAO], 2014). La enfermedad puede presentarse de dos formas clínicas diferentes: la enterocolítica, causada por S. typhimurium y la septicémica por S. cholerasuis (Pastrana et al., 2014).
La forma septicémica se considera la más grave, ésta cursa con fiebre, temperaturas superiores a los 41,7ºC, inapetencia, letargia, cianosis en la parte distal de las extremidades, respiración superficial y tos húmeda, estos signos clínicos aparecen a las 24-36 horas de la infección mientras que la diarrea, por lo general no se presenta hasta 4-5 días después. Suele afectar a cerdos de entre los 2 y 4 meses de edad, con una morbilidad variable, en torno al 10%, y una mortalidad muy alta. También se ha descrito que puede provocar abortos en cerdas (García, 2011).
Esta investigación busca llevar a cabo un análisis de la condición sanitaria de las granjas muestreadas e identificar la presencia de Salmonella spp., específicamente la más perjudicial para lechones destetados (S. choleraesuis), mediante pruebas bioquímicas automatizadas para ayudar posteriormente a los productores a tomar medidas eficaces que logren disminuir la morbilidad y mortalidad en sus granjas, puesto que al identificar la presencia de este patógeno se buscará orientar a los propietarios sobre la implementación de una alimentación adecuada para los lechones y el control de estos tipos de enfermedades por agentes causantes de trastornos gastrointestinales, y de esta manera, realizar un aporte sustancial en cuanto a los riesgos biológicos y epidemiológicos de importancia en salud pública.
Materiales y Métodos
Selección del grupo de estudio La población porcina en Valledupar, según datos de la Coordinación de Vacunación de Peste Porcina Clásica de la Asociación Colombiana de Porcicultores Asoporcicultores para el año 2015, fue aproximadamente 1.854 cerdos, específicamente en la zona de la carrera 4, entre calles 12 a 30 al margen derecho del río Guatapurí se estimó una población de 6000 cerdos, los lechones destetados representaron el 20% de la población total de cerdos de traspatio, siendo un total de 1000 a 1200 animales.
Tamaño de la muestra Se tomaron 90 muestras distribuidas en nueve granjas con producción de traspatio o artesanal a lo largo de la carrera 4, desde la calle 12 hasta la calle 30, dividiéndose en 10 muestras por granja a lechones destetados que presentaron sintomatología de diarrea. El estudio se desarrolló en dos etapas, la primera etapa donde se realizó la toma de muestras en los nueve predios. La segunda etapa fue la de procesamiento de las muestras, ejecutada en los laboratorios de microbiología de la Universidad Popular del Cesar.
Toma de muestras Se solicitó a los dueños de las granjas mediante una carta, la autorización para la toma de muestras en donde se anexó además una encuesta, con el fin de evaluar el estado sanitario de estas granjas en donde se tuvo en cuenta el número de cerdos destetados por granja, suministro de antibióticos y la frecuencia con la que presentaban diarrea. Las muestras de materia fecal se tomaron con un hisopo estéril y se depositaron en un tubo tapa rosca debidamente esterilizado y rotulado.
Procesamiento en laboratorio Aislamiento e identificación de Salmonella cholerasuis Para el aislamiento e identificación de Salmonella spp. se usó el protocolo para salmonelosis propuesto por la Organización Mundial de Salud Animal [OIE] (2008), adaptado y modificado de la siguiente manera: se utilizó hisopado rectal el cual se inoculó en 9 ml de agua peptonada, se incubó a 37˚C a 24 horas (pre-enriquecimiento). Posteriormente se inoculó 1 ml de cada muestra en 10 ml de caldo Rappaport-Vassiliadis y se incubó a 44˚C durante 18 horas (enriquecimiento selectivo).
Se tomó una porción de esta preparación con asa bacteriológica y se sembró por agotamiento en la superficie del agar desoxicolato de lisina y xilosa (XLD), se incubó a 37˚C por 24 horas. A continuación, las colonias sospechosas se repicaron en agar Salmonella – Shigella y agar bismuto sulfito para una mayor selectividad, luego estas se sometieron a identificación con pruebas bioquímicas convencionales (TSI, Urea, VP, SIM, LIA, Citrato, RM).
La confirmación de cepas de serotipo cholerasuis se realizó a través del equipo automatizado Microscan Walkaway 96, el cual se utilizó bajo las instrucciones del fabricante y con la guía de personal autorizado en un laboratorio privado en Valledupar.
Resultados y Discusión
Identificación presuntiva del género Salmonella Para la identificación presuntiva de las cepas de Salmonella se recurrió al uso de pruebas bioquímicas convencionales como: urea, TSI, LIA, CITRATO, VP y RM que según la naturaleza del microorganismo los resultados en las muestras positivas fueron los siguientes: indol (-), RM (+), lisina descarboxilasa (+), glucosa (fermentación) (+) con gas, urea (hidrólisis) (-), citrato (+).
Cuatro de las granjas mostraron resultados presuntivos en las pruebas bioquímicas convencionales, por lo cual se procedió a hacer pases de las muestras en agar nutritivo para su posterior confirmación en el equipo MicroScan WalkAway 96.
Confirmación de Salmonella cholerasuis mediante el equipo automatizado MicroScan WalkAway 96 De las nueve granjas, utilizando el protocolo para la identificación de Salmonella spp., se logró evidenciar que alrededor de un 33% (3/9) resultó positiva para salmonelosis.
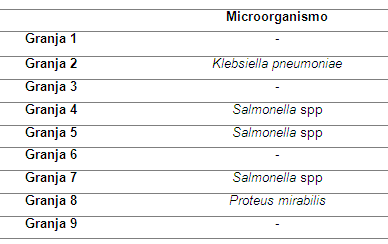
Como se mencionó, de las nueve granjas muestreadas, cuatro de ellas fueron presuntivas para la presencia del género Salmonella; no obstante, una vez procesadas las muestras en el equipo MicroScan Walk Away 96, se logró comprobar que el género Salmonella se encontraba solo en tres. Sin embargo, no fue posible identificar S. cholerasuis, lo que indica que otras subespecies o serotipos de Salmonella diferentes a S. paratyphi A, S. typhi, S. arizona, y S. choleraesuis son las que están causando la enfermedad, ya que este equipo mediante su software puede identificar los grupos mencionados anteriormente (Rochem Biocare & Dade Behring, s.f). Además de las especies identificadas como Salmonella spp. se encontró la presencia de otros microorganismos como Proteus mirabilis y Klebsiella pneumoniae en dos granjas del muestreo (Tabla 1).
Tabla 1. Microorganismos encontrados en el procesamiento de las muestras.
K. pneumoniae se encontró en los cerdos de la granja número dos, en esta granja se encontraron varios animales enfermos y los propietarios reportaron muertes de lechones demasiado frecuentes; aunque los métodos convencionales mostraron que no se trataba del género Salmonella, se llevó al equipo con el fin de determinar el posible microorganismo responsable, resultando ser Klebsiella pneumoniae el cual es un bacilo Gram-negativo, no móvil, y es considerada como la especie de mayor importancia clínica y más estudiada dentro del género Klebsiella (López & Echeverry, 2010).
Proteus mirabilis se encontró en la granja número ocho, su presencia en los resultados se puedo deber a su capacidad de contaminar medios de cultivo, a la similitud de las colonias en el medio Salmonella-Shigella o al resultado de pruebas bioquímicas convencionales relacionadas con Salmonella atípicas, sin embargo P. mirabilis no constituye una amenaza al ser encontrada en las heces de los lechones, debido a que éste hace parte de su flora bacteriana normal (Martínez & Castro, 2004).
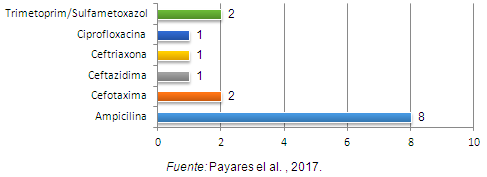
Se probó la resistencia de las cepas de Salmonella spp encontradas con 7 antimicrobianos mediante el equipo automatizado MicroScan: ampicilina, cefotaxima, ceftazidima, ceftriaxona, ciprofloxacina y trimetoprim/sulfametoxazol, y según la CIM, todas las cepas encontradas resultaron sensibles a todos los antimicrobianos testeados, sin embargo la CIM más alta fue para ampicilina (Gráfica 1). Aspecto importante, considerando que en el 89% de las granjas no se aplican medicamentos o tratamientos contra las diarreas.
Gráfica 1. Concentración mínima inhibitoria (CIM) correspondiente a las cepas de Salmonella spp. encontradas en las muestras de la investigación.
Así mismo, Ibar et al. (2009) realizaron un estudio para determinar la prevalencia de Salmonella y sus serovariedades en cerdos de faena, para evaluar sus perfiles de resistencia a los antimicrobianos y para conocer la presencia de integrones de clase 1 como posibles reservorios de resistencia hallando 13 serovariedades de S. enterica, las más prevalentes fueron S. Schwarzengrund, S. Heidelberg, S. subespecie I 6,8:e,h:-, S. Derby y S. Bredeney, encontrando también que la mayoría de las serovariedades fueron sensibles a muchos de los antimicrobianos testeados.
El uso indiscriminado de antibióticos añadidos en la alimentación de los cerdos es uno de los factores más cruciales en la resistencia de los microorganismos como Salmonella spp., tal como lo referencia Calderón et al. (2012) en su estudio sobre la resistencia antimicrobiana de Salmonella spp. a antibióticos utilizados convencionalmente para tratar las infecciones por este microorganismo, por lo que es necesario que los productores eviten utilizar medicación a los animales si no se presentan enfermedades en la producción.
A pesar de que Salmonella spp. no se encontró en grandes porcentajes, solo con la detección de su presencia indica un problema de salud pública, ya que las canales comercializadas no deberían tener indicios de este microorganismo bajo ninguna circunstancia. Se espera poder complementar este estudio con futuras investigaciones que utilicen otros métodos de identificación.
Conclusiones
Se manifestó en el análisis de las condiciones sanitarias de las granjas un déficit bastante alto, puesto que el 100% de ellas no cumplían con las necesidades mínimas para garantizar el bienestar animal, solo teniendo en cuenta la escasa higiene de las porquerizas se considera que estas incrementan las posibilidades de contraer infecciones en cualquiera de las etapas de producción, representando además un riesgo fundamental para los humanos, por lo que es primordial que las autoridades ejerzan un control sanitario para poner un alto a estas prácticas artesanales que amenazan la salud animal y humana. La presencia del patógeno Salmonella indica por sí mismo un problema de salud pública, por lo que esta investigación aporta evidencias para la futura realización de un estudio serológico en estas granjas para determinar el serotipo que está afectando a los lechones y su posible papel zoonótico.
Agradecimientos
A la Universidad Popular del Cesar por apoyar la investigación, específicamente el programa de Microbiología y el Semillero de Investigación EPIVETE, a los porcicultores por facilitar la toma de muestras en sus animales y hacer parte del proyecto.
Referencias bibliográficas
Araque, H. Sistemas de producción de cerdos. Instituto y Departamento de producción animal. 2009. Maracay: Venezuela. En: Link. Accesado en: 03/05/2017.
Artunduaga, C.M.; Gómez, F.E. Salud animal: un enfoque integral. Colombia: Bogotá. Editorial Universidad Santo Tomas [USTA]; 2013.
Blumetto, O. Caracterización de sistemas de producción porcino y vacuno de engorde en Uruguay. 2012. Disponible en: Link. Accesado en: 11/04/2017.
Bustos, P.; Segura, C. Incidencia de Salmonella spp. y E. coli en tres granjas porcícolas ubicadas en los municipios de Fómeque y Sibaté. 2005. Disponible en: Link. Accesado en: 11/04/2017.
Calderón, L.; Motta, P.; Cerón, M.; Chimonja, F. Resistencia de Salmonella a los antimicrobianos convencionales para su tratamiento. Revista CES Medicina Veterinaria y Zootecnia. v.7, n.1, p. 116-129. 2012.
Campabadal, C. Guía técnica para alimentación de cerdos. 2009. Disponible en: http://www.mag.go.cr/bibliotecavirtual/a00144.pdf Accesado en: 13/04/2017.
Cano, J.; Márquez, Z.; Fuentes D.; Zannin, L.; Utrera, V.; Cordero, F.; Sogbe, E. Diagnóstico y control de la Salmonelosis porcina. Maracay: Venezuela. 2007. Disponible en: Link. Accesado en: 20/04/2017.
Chiu, C.H.; Hui-Do, L.H.; & Chu, C. Salmonella enterica Serotype choleraesuis: Epidemiology, Pathogenesis, Clinical Disease, and Treatment. ClinMicrobiolRev. V.17, n.2, p. 311-322, 2004.
Corporación Colombiana de Investigación Agropecuaria [CORPOICA]. Manual de porcicultura. Capacitación tecnológica para pequeños productores con subproductos de la caña en el departamento de Cundinamarca. 2003. Disponible en: Link. Accesado en: 20/04/2017.
Corrales, L.; Peña, V.; Caicedo, D. Identificación de Salmonella y Escherichia coli en manos y guantes de manipuladores en planta de sacrificio y faenado de un municipio de Cundinamarca. Revista Universidad Colegio Mayor. v.6, n.9, 2008. Disponible en: Link. Accesado en: 24/05/2017.
Figueroa, I.M.; Verdugo, A. Mecanismos moleculares de patogenicidad de Salmonella spp. Revista Latinoamericana de Microbiología. v.47, n.1-2, p. 25-42, 2005.
Flores, R. Epizootiología de la salmonelosis en bovinos, porcinos y aves. 1981. Disponible en: Link. Accesado en: 20/06/2017.
García, C. Salmonelosis porcina en España: prevalencia, factores de riesgo y resistencia antimicrobiana. 2011. Disponible en: Link. Accesado en: 30/06/2017.
Gómez, E.; Hamilton, M.; Retamal M.; Urcelay, V. Identificación de cepas de Salmonella spp. resistentes a antimicrobianos, y factores de riesgo para su circulación, en aves y cerdos mantenidos en sistemas productivos de traspatio de la región del libertador general Bernardo O’Higgins, Chile. International Journal of Applied Science and Technology. v.5, n.2, p.62-67, 2015.
Henao, J.; Ramírez, E.; Rondón, I. Análisis de las Buenas Prácticas de Producción en granjas porcícolas del departamento del Tolima y factores de riesgo asociados a la presencia de Salmonella spp. Revista CES Medicina Veterinaria y Zootecnia. v.7, n.2, p.11-20, 2012.
Ibar, M.; Vigo, G.; Piñeyro, P.; Caffer, M.; Quiroga, P.; Perfumo, C.; et al. Serovars of Salmonella enterica subspecies enterica and its antimicrobial resistance in slaughterhouse pigs. Revista Argentina de Microbiología. v.41, n.3, p.156-162, 2009.
Instituto Colombiano Agropecuario [ICA]. Resolución 2640 de 2007. Diario Oficial No. 46.768 de 1 de octubre de 2007. 2007. Disponible en: Link. Accesado en: 11/06/2017.
Instituto Nicaragüense de Tecnología Agropecuaria [INTA]. Principales Enfermedades de los Cerdos. 2010. Disponible en: Link. Accesado en: 21/07/2017.
López, J.; Echeverri, L.K. pneumoniae: ¿la nueva “superbacteria”? Patogenicidad, epidemiología y mecanismos de resistencia. Revista IATREIA. v.2, n.2, p.157-165, 2010.
Martínez, O.; Castro, E. Flora fecal normal en lechones sin enfermedad aparente. REDVET. Revista Electrónica de Veterinaria. 2004. Link. Accesado en: 24/06/2017.
Mejía, W.J. Epidemiología de la salmonelosis porcina en granjas de Cataluña y determinación de los factores de riesgo de la infección. 2003. Disponible en: Link. Accesado en: 29/04/2017.
Ministerio de Agricultura y Desarrollo Rural. Decreto Número 930 De 2002. 2002. Disponible en: Link. Accesado en: 15/04/2017.
Ministerio del Medio Ambiente, Sociedad de Agricultores de Colombia y Asociación Colombiana de Porcicultores. Guía ambiental para el subsector porcícola. 2002. Disponible en: Link. Accesado en: 11/07/2017.
Oliveira, L.G. Infecção experimental por Salmonella entérica subspécie entérica sorotipo Panamá e tentativa de transmissão naso-nasal em leitões desmamados. 2008. Disponible en: Link. Accesado en: 13/08/2017.
Organización de las Naciones Unidas para la Agricultura y la Alimentación [FAO]. Cerdos y la producción animal. 2014. Disponible en: Link. Accesado en: 21/01/2017.
Organización Mundial de Salud Animal [OIE]. Salmonelosis. Manual de la OIE sobre animales terrestres. 2008. Disponible en: Link. Accesado en: 09/12/2016.
Padilla, M. Manual de Porcicultura. San José: Costa Rica. 2007. Disponible en: Link. Accesado en: 25/06/2017.
Pastrana, A.; Mogollón, J.; Rincón, M. La salmonelosis porcina y su importancia en la cadena de producción. Revista Argentina de Producción Animal. 2014. Disponible en: Link. Accesado en: 11/08/2017.
Pineda, Y. La salmonelosis porcina. Revista Digital CENIAP HOY. Maracay, Aragua, Venezuela. 2005. Disponible en: Link. Accesado en: 30/07/2017.
Rochem Biocare.; Dade Behring. Manual de Entrenamiento Microscan Labpro, s.f.
Rodríguez, D.; Suarez, M. Salmonella spp. in the pork supply chain: a risk approach. Revista Colombiana de Ciencias Pecuarias RCCP. v.27, p.65-75, 2014.
Rodríguez, D.; Torres, F.; Gutiérrez, E.; López, M.; Martínez, M.; Carrascal, A. Salmonella typhimurium determination in compost artificially inoculated in a lettuce crop. Acta biol. Colomb. v.13, n.3, p.61 – 74, 2008.
Sánchez, M.; Cardona, N. Validation of a PCR for diagnosis of typhoid fever and salmonellosis by amplification of the hilA gene in clinical samples from Colombian patients. Journal of Medical Microbiology. v.53, n.9, p. 875-878, 2004.
Vargas, J.; Clavo, N.; Mattar, S. Detección de Escherichia coli 0157 H7 y Salmonella spp en cerdos en del departamento de Córdoba. Revista MVZ Córdoba. v.9, n.1, p.386-392, 2004.
Vásquez, J.E. Uso de probióticos en la alimentación con suero de leche en cerdos al destete. Soledad de Graciano Sánchez: México. 2013. Disponible en: Link Accesado en: 04/02/2017.
Vega, M. Susceptibilidad antimicrobiana de serogrupos de Salmonella spp. aislados de animales domésticos en la provincia de Villa Clara, Cuba. 2012. Disponible en: Link. Accesado en: 05/02/2017.
Zabaleta, G. Evaluación de susceptibilidad antimicrobiana de Salmonella spp., aisladas en la cadena cárnica porcina en tres regiones del país. Bogotá: Colombia. 2014. Disponible en: Link Accesado en: 21/03/2017.
Como citar: Payares-Ramírez, K.; Cañate-González, A.; Polo-Gutiérrez, W.; Prada-Herrera, J.; Esquea-Larios, K. Salmonelosis en lechones destetados de granjas de traspatio en Valledupar, Colombia. Revista Veterinaria y Zootecnia, v. 13, n. 1, p. 57-67, 2019. DOI: 10.17151/vetzo.2019.13.1.4 http://vetzootec.ucaldas.edu.co/index.php/component/content/article?id=265
Esta obra está bajo una Licencia de Creative Commons Reconocimiento CC BY
|